近日,北京林业大学林木分子育种团队联合中国科学院北京基因组研究所(国家生物信息中心)开发了多年生木本植物遗传信息数据库PPGR (https://ngdc.cncb.ac.cn/ppgr/),该研究成果以“PPGR: a comprehensive perennial plant genomes and regulation database”为题被《核酸研究》(Nucleic Acids Research)(ICR分区一区,IF="14.9)收录并发表。
多年生木本植物是重要的植物类群,是木材、精油、松脂等特殊代谢物的重要来源,对整个生态系统、工业和医药产业更有着巨大的贡献。因此,精确解析多年生木本植物木材形成、抗性调控、休眠机制、次生代谢调控等重要生物学过程关键基因,建立系统的遗传调控网络,对于阐明该类群植物重要性状生物学基础、分子育种和种质创新具有重要的意义。
近年来,随着多年生木本植物的基因组和功能研究的深入,大量的数据不断产生。然而,这些数据的收集、存储管理、功能挖掘和开发利用一直是该领域发展的核心问题。为了解决这些问题,PPGR在多年生木本植物数据标准化的基础上,提供了多组学大数据存储、分析、共享和可视化服务。PPGR的开发实现了多年生木本植物组学资源的系统管理和高效利用,为多年生木本植物遗传改良、功能基因组学研究、分子育种提供了新的重要遗传资源和有力工具。
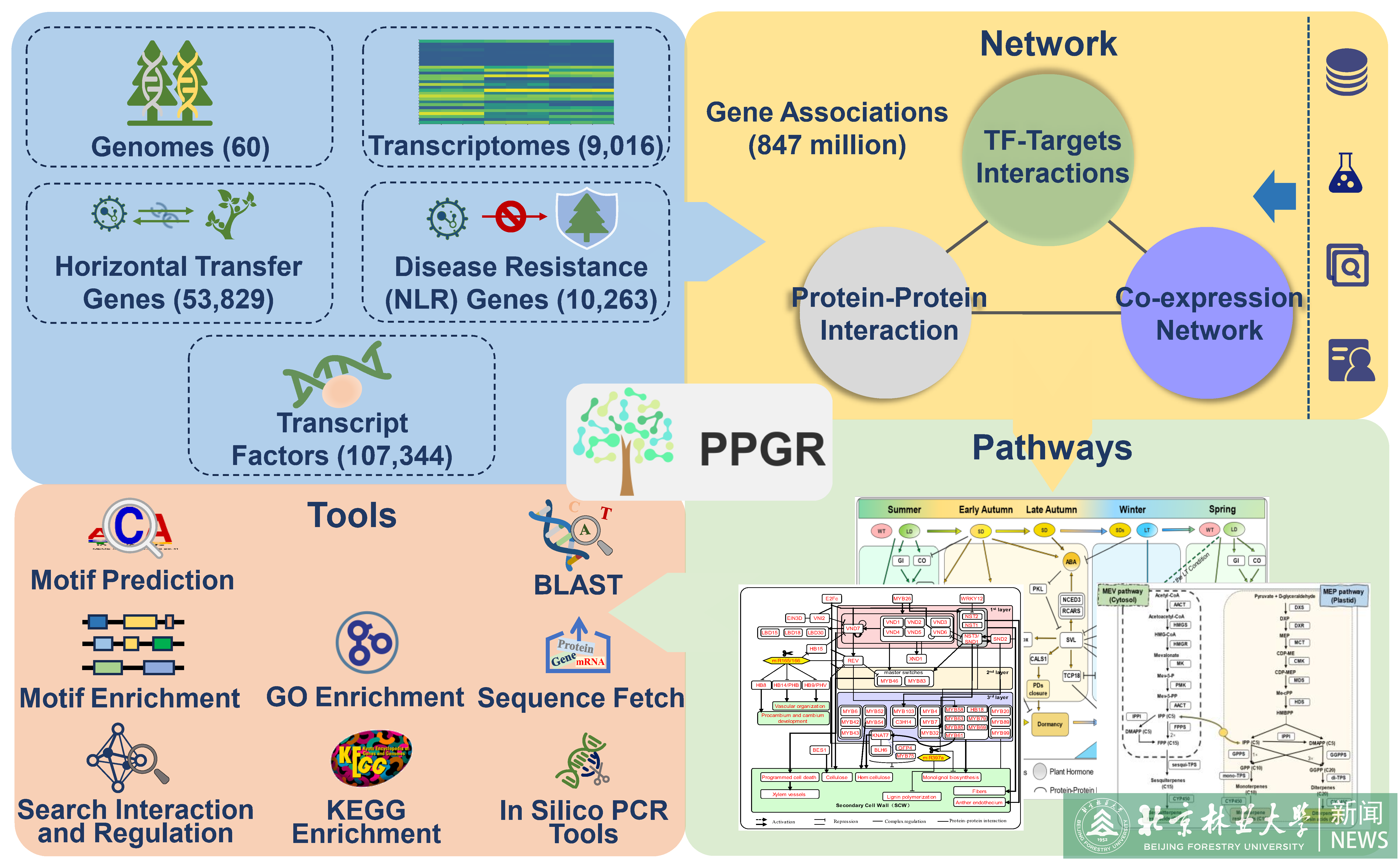
PPGR整合了毛果杨、油松、大桉、银杏等60种木本植物的基因组数据,应用标准化流程分析了9,016个转录组数据集,PPGR计算了107,344个转录因子、10,263个抗病 (NLR) 基因、以及53,829个水平转移基因、8.47亿条基因互作对用来构建多维基因调控网络,重绘了多条木本植物重要代谢通路(木材形成、季节性休眠、萜烯生物合成和叶片衰老),其中包涵了87,372个通路候选基因。另外PPGR还提供了相应模块供用户浏览分析多组学数据,具备较强的实用性和交互性,并且提供了“一站式”分析结果的在线集成工具以方便用户对基因集合进行功能富集分析 (GO、KEGG)、调控网络检索、序列分析(启动子序列motif 预测与富集) 和BLAST。
PPGR: 多年生木本植物遗传信息数据库
PPGR是一个全面的多年生木本植物资源库,用户可以通过基因名称或基因索引快速获取感兴趣基因的位置、结构、功能域、代谢通路、同源基因、互作基因、调控通路、表达模式等相关多组学信息。在多维基因调控网络构建过程中,PPGR采用了文本智能挖掘、文献数据人工审编、互作预测算法等多种方式对蛋白-蛋白、转录因子-靶标、共表达网络在内的多种信息进行高效整合并预测。除此之外,基于遗传调控网络的分子育种模块正在开发,近期将应用于该数据库。
截止2024年3月,PPGR已成功收录了众多与多年生植物基因组和调控相关的数据,这一宝贵的资源无疑将加速多年生植物基因组和调控研究领域的发展。随着PPGR数据库的推出,预计将为林木基因组学和基因调控研究领域带来更多的科研突破和发现。
北京林业大学生物学院硕士研究生杨森,中国科学院基因组研究所博士研究生宗文婷为论文第一作者;北京林业大学生物学院谢剑波教授,中国科学院北京基因组研究所李茹姣研究员,中国科学院北京基因组研究所鲍一明研究员为通讯作者。研究工作得到国家重点研发计划、国家自然科学基金优秀青年基金、国家林草局青年拔尖人才等项目的联合资助。
论文链接:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkad963/7337612?searchresult="1