近日,生物学院高宏波教授课题组在国际著名植物学期刊Plant Physiology上在线发表了题为“Sequence variations affect the 5’ splice site selection of plant introns”的研究论文,对内含子5’ss的序列识别机制进行了较为深入的分析研究,揭示了一些不为人知的机制和规律。
真核生物中的基因大多是含有内含子的断裂基因。转录过程中产生的前体信使RNA(mRNA)要经过剪接体的剪接去除内含子,才能形成成熟的mRNA。RNA剪接的第一步是U1 snRNP识别5’端剪接位点(5’ splice site,5’ss)。该过程是一直被认为是通过碱基互补配对实现的,以AG/GUAAG为最佳组合,一些与之相似的序列也可以被识别,但是+1和+2位的G和U是极其保守的。5’ss的序列变异会导致替代5’ss的出现,这不仅影响到基因的编码序列和功能,也会影响到基因注释的准确性。
课题组在分析数据库中叶绿体分裂关键蛋白FtsZ1的序列的时候发现有些植物在FtsZ1的序列保守区有插入和缺失,进一步的分析发现这是由于在这些植物中的一个内含子5’ss是非典型的GC位点,而计算机因为选择了常见的GU位点,导致基因的内含子和外显子预测错误。对拟南芥、水稻、玉米、棉花和月季等多种植物的基因组分析发现,约1%左右的内含子是以GC起始,但这也涉及到上千个基因。
通过对剪接位点不同的碱基进行突变,观察其对剪接位点的识别和剪接效率的影响,并结合分子动力学模拟,课题组发现将+2位突变为A或G时,5’ss的剪接受到严重的影响。因为A和G为嘌呤,大于为嘧啶的C和T,说明空间位阻对5’ss的识别也有重要影响。类似的情形在其他位点的突变实验中也存在,进一步支持了上述观点。通过比较拟南芥基因组中GC内含子和GU内含子的5’ss序列,课题组发现GC内含子对识别序列的保守性要求更高(图1)。这些结果说明碱基配对和空间位阻都在内含子5’ss序列的识别过程中发挥重要作用。
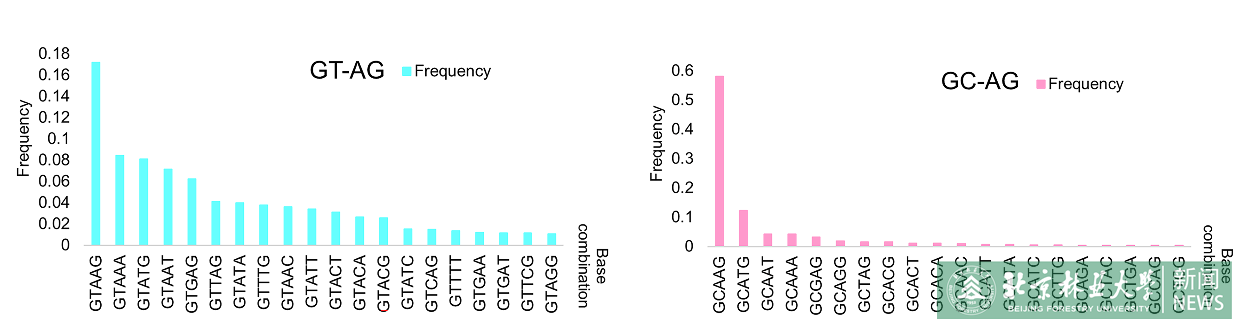
图1 拟南芥基因组中GT-AG和GC-AG内含子5’ss的 +3到+5位不同序列的频率(前20个组合的频率)
在内含子的5’ss附近经常会有隐藏的5’ss,它们通常不会被识别。在主要的5’ss突变后,隐藏的5’ss会被识别,这会造成基因的错误剪接。研究发现,如果两个接近的5’ss在竞争力有较大差异的情况下,剪接体只会识别主要的5’ss,这是大多数内含子剪接的情形。在两个接近的5’ss竞争力差异不大的情况下,会导致替代5’ss的出现,进而会导致不同剪接异构体的产生。如果一个内含子的5’ss较弱,内含子则不容易被剪接从而产生滞留。
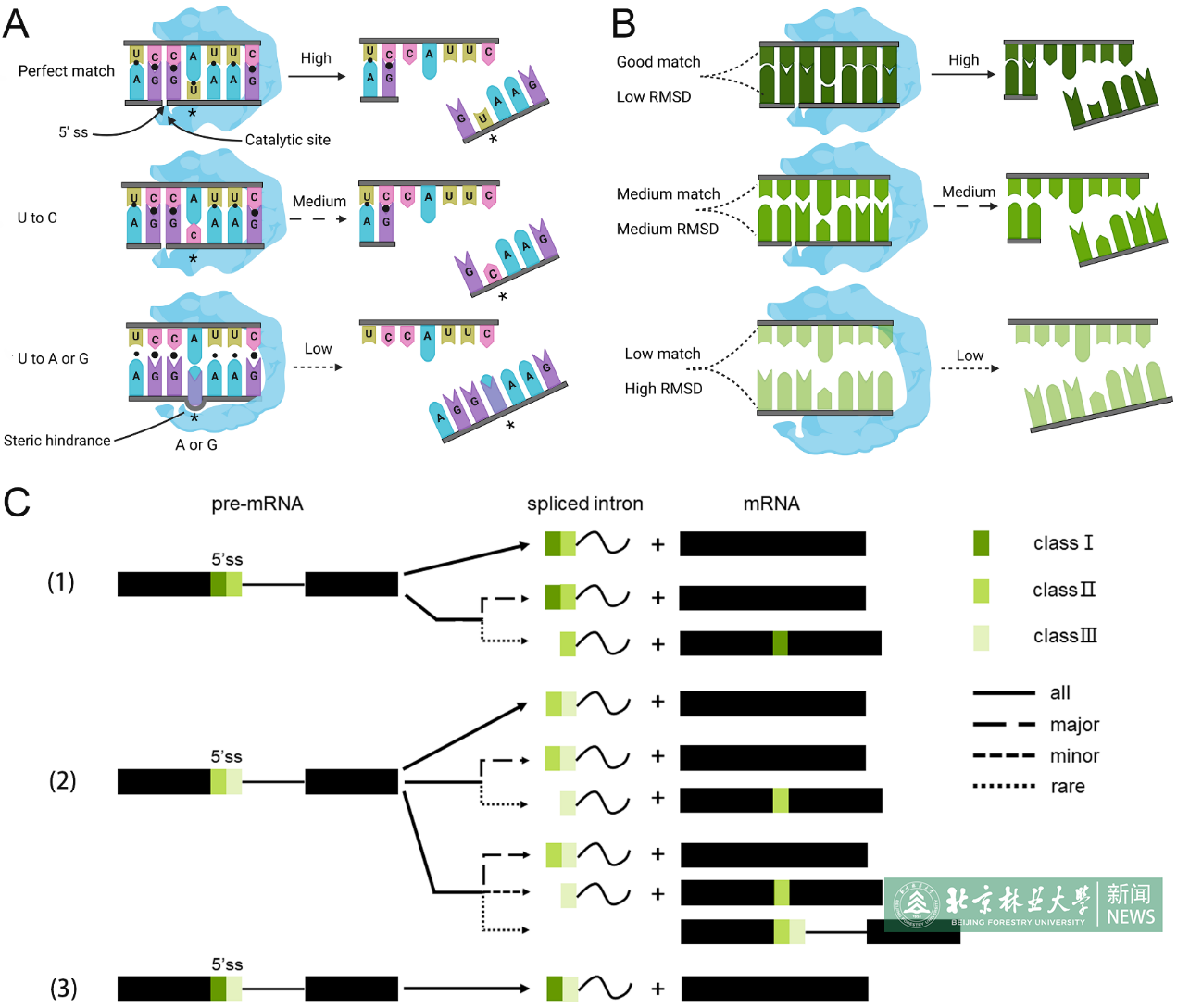
图2 序列变异影响内含子5’ss识别和选择的模式图
基于论文中的研究发现,作者提出了一个较为详细的内含子5’ss识别和选择的模型(图2)。该项工作不仅加深了人们对内含子5’ss的序列识别和选择机制的认识,还有利于提高基因注释和预测的准确性。
生物学院高宏波教授为该论文的通讯作者,博士研究生程文真为该论文的第一作者。博士研究生洪琮浩在生物信息学分析方面做出了重要贡献。该研究得到了中央高校基本科研业务费专项资金(2022BLRD14)和国家自然科学基金的资助 (32070696)。
论文链接:https://academic.oup.com/plphys/advance-article/doi/10.1093/plphys/kiad375/7216941